Forschungs- und Innovationsprojekt
Digi-Test: Neue Direktnachweise für Kartoffelviren und -bakterien

Entwicklung einer multiplexen digitalen DiRT-PCR für die Kartoffelknollentestung auf Viren und Pathogene der Schwarzbeinigkeit und bakteriellen Welke aus demselben Probenmaterial
Gesundes Kartoffelsaatgut ist essentiell für eine langfristige Versorgung der Bevölkerung mit Kartoffeln in hoher Qualität. Es ist daher wichtig, relativ früh und zuverlässig den Krankheitsbesatz festzustellen. Im Projekt Digi-Test untersuchen wir, wie Kartoffelproben aus dem Feld direkt, sensitiv und zeitsparend im Labor auf viele Viren und Bakterien gleichzeitig getestet werden können.
Ziel des Projektes ist die Entwicklung eines hochmodernen nucleinsäurebasierten Testverfahrens, das mehrere Viren und Bakterien direkt auf dem verdünnten Saft der Kartoffelknolle nachweist. Dieses kann bisherige, zeitintensivere Standard-Verfahren ganz oder teilweise ersetzen.
Ziele
Entwicklung eines geeigneten Nachweis-Verfahrens das die folgende Anforderungen erfüllt
- nucleinsäurebasierter Nachweis von Viren und Bakterien (real-time quantitative Polymerase Kettenreaktion)
- direkter Nachweis aus dem Press-Saft der dormanten Kartoffelknolle (DiRT-qPCR)
- gleichzeitiger Nachweis mehrerer Viren und Bakterien in einer Reaktion (Multiplex-DiRT-qPCR)
- Möglichkeit der gleichzeitigen Testung mehrerer Einzelproben (Pool-Proben)
Methoden
Bedeutung und Testung der Kartoffelviren
Auf Grund dessen wurden in den letzten Jahren bereits die sensitivere und schnellere Multiplex-qRT-PCR (Multiplex quantitative real-time Polymerase-Kettenreaktion) entwickelt, die mehrere Viren in einer Reaktion testet. Die bisher etablierten Reaktionen erfordern allerdings eine teure und zeitraubende Aufreinigung der Viren-RNA. Für ein Hochdurchsatzverfahren in der gängigen Virentestung muss deshalb nach effizienteren Möglichkeiten gesucht werden.
Phänotypische Ausprägung mit Viren infizierter Kartoffelpflanzen
Bedeutung und Testung der Schwarzbeinigkeitsbakterien auf Kartoffeln
Latenter Befall mit Schwarzbeinigkeitsbakterien ist auf dem Feld nicht zu erkennen. Bei entsprechenden Witterungs- und Lagerbedingungen können sich die Erreger vermehren und zu großen Schäden führen. Demzufolge wird der Bedarf immer höher, das Pflanzgut vor der Ausbringung auf die häufigsten und virulentesten Bakterienarten zu testen. Aktuell kann auf mehrere Bakterien gleichzeitig in der konventionellen PCR getestet werden. Jedoch sind hier wieder aufwendige DNA-Extraktionen und Elektrophorese-Gelanalysen notwendig.
Ergebnisse
Aktueller Stand und Projektplan
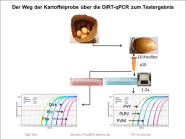 Zoombild vorhanden
Zoombild vorhanden
Schematischer Weg der Kartoffelknollenprobe vom Kartoffelsack bis um fertigen Testergebnis
Abkürzungen für die Erreger im Diagramm: Dick=Dickeya, Pcc=Pectobacterium carotovorum carotovorum, Pba=Pectobacterium atrosepticum, PVY und PVM=Potato Virus Y und M, PLRV=Potato Leaf Roll Virus=Blattrollvirus.
Projektfortschritt und Überführung in die Routinetestung
In dieser Testsaison 2020/21 wurde die Validierung in der Pooltestung abgeschlossen. Hier wurde die multiplexe real-time DiRT-PCR mit einer Extraktions-PCR-Methode und mit ELISA verglichen. Die Übereinstimmung ist diesmal durchweg sehr gut.
Projektinformation
Laufzeit: 01.06.2016 bis 30.11.2021
Kostenträger: Bayerisches Staatsministerium für Ernährung, Landwirtschaft und Forsten (StMELF)
Pflanzkartoffel-Fördergemeinschaft des Landesverbandes der Saatkartoffel-Erzeugervereinigungen in Bayern e.V.
Projektleitung: Gerda Bauch (IPZ6a), Adolf Kellermann (IPZ3a)
Projektbearbeitung: Dr. Mirjam Prinz (IPZ6a und 3a)
Technische Assistenz: Madlen Ludwig (IPZ6a)
Förderkennzeichen: A/18/11
Kontakt
Dr. Mirjam Prinz
Tel.: 08161 71-3636
E-Mail: Mirjam.Prinz@lfl.bayern.de












