Innovations- und Forschungsprojekt
Von Südostasien nach Bayern: Soja heimisch machen
Verbesserung der heimischen Soja: Entwicklung und Etablierung markergestützter Kreuzungspläne zur merkmalsbezogenen, gezielten Kombination von Genbankmaterial und hochwertigem Zuchtmaterial anderer geografischer Herkünfte.
Im Rahmen der Eiweißinitiative Bayerns und der Biodiversitätsstrategie wurde an der Landesanstalt für Landwirtschaft (LfL) seit 2014 ein umfangreiches Sortiment von Genbankakzessionen aus Deutschland, der Schweiz, Frankreich, Osteuropa, Kanada, China, Korea und Japan angebaut und erstmalig genotypisch und phänotypisch unter heimischen Anbaubedingungen umfassend bewertet. Stammbaumanalysen basierend auf mehreren tausend DNA-Markern haben gezeigt, dass die derzeit im Praxisanbau befindlichen Sojasorten zwar gute Ertragsleistungen haben, genetisch jedoch wenig Variation zeigen. Demgegenüber steht eine große vorhandene genetische Diversität im geprüften Genbankmaterial – dieses zwar mit geringerem Ertragsniveau, aber einem großen Potential für die Verbesserung wesentlicher Merkmale. Das Genbankmaterial bietet damit gute Möglichkeiten zur gezielten Erweiterung des Sortengenpools im Hinblick auf eine bessere Anpassung der Sojapflanzen an heimische Anbaubedingungen. Mit der Etablierung markerbasierter Kreuzungspläne kann mittels Präzisionszucht (SMART breeding) diese enge genetische Fixierung gezielt aufgebrochen und neues Pre-breeding Material für die bayerische Sojazüchtung bereitgestellt werden.
Ausgangssituation
Der Anbau von Soja beginnt mit der Aussaat im April/Mai und endet mit der Ernte reifer brauner Hülsen im September/Oktober. Unter süddeutschen Klimabedingungen ist es für die aus Südostasien stammende Soja schwer, rechtzeitig vor dem Winter reife Hülsen zu produzieren. International existieren bei Soja 13 Reifegruppen MG 000 (sehr frühreif) bis MG X (tropisch), von denen aufgrund der erforderlichen rechtzeitigen Abreife nur die frühen Reifegruppen MG 000 bis MG 00 für Bayern relevant sind. Sojapflanzen dieser Reifegruppen sind in der Lage in der heimischen Vegetationsperiode reife Sojasamen zu liefern. Allerdings finden diese frühen Reifegruppen in der internationalen Forschung bislang nur wenig Berücksichtigung, obgleich die Bedeutung aufgrund ihrer begünstigten Abreife bei Sommertrockenheit auf breiter Ebene zunimmt.
Forschungsziel
Ziel des Vorhabens ist die phänotypische und genotypische Charakterisierung ausgewählter Genbankakzessionen und Sorten für die Entwicklung molekulargenetischer DNA-Marker (Assoziationsstudie) zur Etablierung markergestützter Kreuzungspläne (SMART breeding) bei Soja. Im Mittelpunkt stehen Untersuchungen im Feld und im Labor für die zentralen Merkmale Blühzeitpunkt, Reife, Terminierung und Kühletoleranz sowie Qualitätsparameter im Sojasamen (Aminosäurezusammensetzung, Protein- und Ölgehalt).
Die natürliche Vielfalt in Feld und Labor aufzeigen
Eine Auswahl von 117 Genbankakzessionen und Sorten wurde als Materialset zusammengestellt und wird in Feldversuchen an verschiedenen bayerischen Standorten phänotypisch beschrieben. Dazu werden Daten für die Merkmale Blühzeitpunkt, Blütenfarbe, Terminierung und Reifezeitpunkt erhoben.
Im Besonderen wird mittels eines Klimakammerversuchs ein indirektes Merkmal zur Erfassung und Bewertung der Kühletoleranz etabliert. Hierzu wird der Einfluss von Kühlestress im Keimlings- und Sämlingsstadium unter kontrollierten Bedingungen in der Klimakammer untersucht. Für die Bestimmung der Aminosäurezusammensetzung sowie des Protein- und Ölgehalts werden NIRS-Messungen im LKV in Grub durchgeführt. Im DNA-Labor werden mittels moderner, molekulargenetischer Methoden i) Kandidatengene für die oben genannten Merkmale im Materialset resequenziert, um natürlich vorkommende DNA Varianten (Polymorphismen) aufzudecken, ii) in internationalen Publikationen beschriebene Polymorphismen auf ihre Diversität im Materialset untersucht und iii) mittels illumina® Genotypisierung genomweite Daten zu Einzelnukleotidpolymorphismen (single nucleotide polymorphism = SNP) erhoben.
Verknüpfung von Feld und Labor - Entwicklung von Selektionsmarkern
Sämtliche phänotypische und molekulargenetische Daten werden einer genomweiten Assoziationsstudie zugeführt, um eine Polymorphismus-Merkmal-Wechselwirkung zu prüfen. Finden sich statistisch signifikante DNA Polymorphismen, die einen putativen Einfluss auf ein oder mehrere geprüfte Merkmale haben, werden diese als umweltunabhängige molekulare Marker für die Selektion von Kreuzungspartnern in Züchtungsprogrammen herangezogen.
Vom Labor aufs Feld: Entwicklung SNP-basierter Selektionschips
Für die praxisorientierte Umsetzung der entwickelten molekularen Marker werden SNP-basierte Selektionschips zusammengestellt, die auf der Fluidigm EP1™ Plattform zur Genotypisierung genutzt werden. Hierfür werden mehrere SNP-Marker mit signifikanter Assoziation zu den Zuchtmerkmalen Blühzeitpunkt, Reife, Terminierung, Kühletoleranz, Aminosäurezusammensetzung sowie Protein- und Fettgehalt für Selektionsprogramme ausgewählt. Erste Selektionschips zur Kontrolle erfolgreicher Kreuzungen, Aufklärung von Saatgutvermischung und Auswahl frühreifer Sojaakzessionen wurden bereits erfolgreich entwickelt. Mit der Fluidigm-Nanofluidtechnik wurde eine schnelle, kostengünstige und praxisnahe Genotypisierungsplattform im Genomanalyselabor an der LfL etabliert. Mit Hilfe entwickelter Selektionschips für die Fluidigmplattform können zukünftige Kreuzungsprogramme in der Sojazüchtung markergestützt begleitet und beschleunigt werden.
Projektergebnisse
Heimisches Soja gilt als Hoffnungsträger der Landwirtschaft und nirgends in Deutschland werden mehr Sojabohnen angebaut als in Bayern. Die Sortenzüchtung musste aufgrund der Nachfrage schnell nachziehen, da die verfügbaren Sojabohnensorten genetisch sehr eng und ihre Qualität, Ertragsstabilität und Proteinerträge noch unzureichend für die Markterfordernisse sind. Besonderes Augenmerk ist auf die rechtzeitige Abreife und der hiermit verbundenen Ertragssicherheit unter hiesigen klimatischen Bedingungen zu legen. Späte Reifegruppen dreschen zwar besser, aber ihre Ab-/Druschreife ist in vielen Anbaujahren nicht gesichert. Die Entwicklung effektiver Züchtungsstrategien zur Verbesserung der Anbaueignung und des Proteinertrags ist deshalb besonders gefragt. Die markergestützte Selektion dient, wie im Projekt gezeigt, als zentrale Methode zur Unterstützung der Pflanzenzüchtung beim Erreichen dieser Zuchtziele.
Im Projekt konnten wesentliche Ergebnisse zur Züchtung auf Frühzeitigkeit und damit zur Verbesserung frühreifer Genotypen der heimischen Sojabohne (Glycine max) erzielt werden. Im Vordergrund standen die Markerentwicklungen zur Sortendifferenzierung, Kreuzungskontrolle und zur Selektion von Sojalinien mit verbesserten Eigenschaften bezüglich früher Blüte, früher Reife und Kühlestresstoleranz. Diese Marker werden nun in der genetischen Analyse von Sojabohnenmaterial eingesetzt, um Linien mit züchtungsrelevanten Merkmalskombinationen zu selektieren und Rückschlüsse auf die genetische Verwandtschaft abzuleiten (Stammbaumanalysen). Diese ist notwendig, denn die Sojabohne (Glycine max) besitzt 20 Chromosomen (Gm01 - Gm20) mit über 46.000 Genen. Zur Unterstützung der Selektion in klassischen Züchtungsprogrammen konnten über das vorliegende Forschungsprojekt nun drei Markerarrays basierend auf der Fluidigm® SNP Genotypisierungstechnologie entwickelt werden:
FluidigmSoja-Diversity
ein Markerarray mit 93 SNP-Markern der zur Sortenidentifikation, für die Kontrolle von Kreuzungserfolgen
- zur Bestimmung der genetischen Diversität und
- für die Feststellung von Saatgutvermischungen herangezogen werden kann.
FluidigmSoja-EarlyFlower
Ein Markerarray mit 24 SNP-Markern der zur gezielten Selektion von frühblühenden und frühreifen Genotypen dient. Mit diesem SNP-Array „EarlyFlower“ kann zukünftig die Selektion auf photoperiodisch insensitive Genotypen bei Soja markergestützt erfolgen und zusammen mit den Haplotypen des Kandidatengens GmFT5a 40% der genetischen Variation für Blühzeitpunkt und 30% der genetischen Variation für Reifezeitpunkt der Hülsen erklären. Der Array stellt damit eine wichtige Selektionsunterstützung für die Aufstellung der Kreuzungspläne für die Entwicklung frühreifer Sojastämme dar.
FluidigmSoja-Cold
Ein Markerarray mit 24 SNP-Markern zur SNP-basierten Selektion auf kühlestresstolerante Genotypen und dem Ziel, einen früheren Saattermin zu erreichen. Die Eignung der SNP-Marker wird derzeit bioinformatisch und technisch geprüft und steht zur Selektion auf kühlestresstolerante Sojaakzessionen zur Verfügung.
Mit allen drei Markerarrays basierend auf der Fluidigm-Nanofluidtechnik wurde an der LfL eine schnelle, flexible und praxisnahe Methode zur Genotypiersung bei Sojabohne etabliert. Sie dient nun der frühzeitigen und umweltunabhängigen Selektion von ungeprüftem Züchtungsmaterial für die Sojabohnenzüchtung.

Übersicht der Projektarbeiten
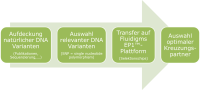
Etablierung von Selektionschips
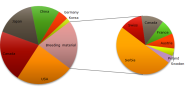
Herkunft der 117 Sojaakzessionen
Projektinformation
Projektleitung: Dr. G. Schweizer, Dr. B. Büttner
Projektbearbeitung: Dr. G. Schwertfirm
Kooperationspartener: LfL, Institut für Pflanzenbau und Pflanzenzüchtung, Pflanzenbausysteme und Züchtungsforschung bei Mais
Projektlaufzeit: 01.09.2017 - 31.08.2020
Finanzierung: Bayerisches Staatsministerium für Ernährung, Landwirtschaft und Forsten
Förderkennzeichen: E/17/01










